登陆有奖并可浏览互动!
您需要 登录 才可以下载或查看,没有账号?立即注册


×
组蛋白翻译后修饰及研究方法介绍1 组蛋白修饰基础知识介绍 // 真核生物中染色质的基本结构单位是核小体,其由组蛋白八聚体和缠绕在外的DNA组成。每个核小体又是由两个亚基构成的,而每个亚基包括H2A、H2B、H3和H4这四个组蛋白。组蛋白尾部的蛋白结构域带正电荷,与带负电荷的DNA相结合。其最N末端部分不参与核小体组装,而是从核心结构中突出(图1)[1],更适合与相邻环境进行相互作用,因此易受翻译后修饰(PTM)的影响。在四种不同的组蛋白质中,组蛋白H3和H4的尾部比H2A和H2B更容易发生PTM[2]。 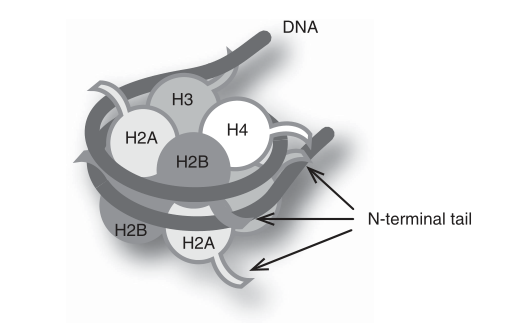 图1 核小体的结构
// 常见的组蛋白PTM包括甲基化、乙酰化、磷酸化、泛素化、ADP核糖基化等(图2)。这些修饰方式可以多种组合形式出现,发挥不同的生物学功能。组蛋白发生修饰后会使染色质的构象等发生变化,从而影响了DNA与组蛋白之间的相互作用,进而调控基因的转录表达。可以说,组蛋白翻译后修饰与DNA甲基化是基因和表观调控的基本手段,也是细胞和发育过程的基础[2]。下面以组蛋白乙酰化和甲基化为例做详细介绍。 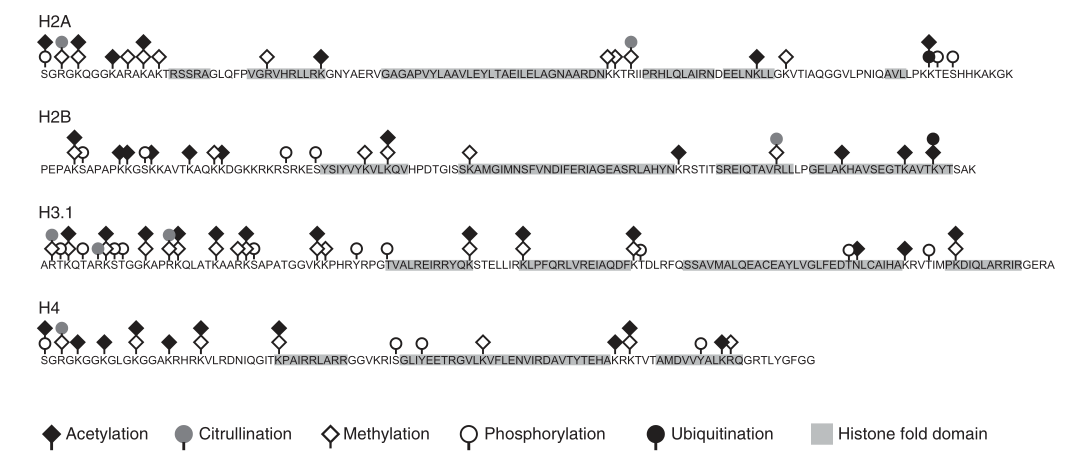 图2 核心组蛋白常见的翻译后修饰类型
2 组蛋白乙酰化修饰 //
组蛋白乙酰化是一种常见的组蛋白PTM,其在真核细胞基因表达的表观遗传调控中发挥重要作用。与其它PTM一样,乙酰化也是一个可逆的过程,且组蛋白的乙酰化水平是由组蛋白乙酰化酶和去乙酰化酶共同决定的(图3)。在生物体内,乙酰化和去乙酰化这两个过程呈动态平衡,一起来调控基因的转录表达[5]。一般情况下,组蛋白乙酰化会激活基因表达,而去乙酰化则会抑制基因表达[5]。
组蛋白的乙酰化经常发生在组蛋白N基末端特定的赖氨酸残基上,比如H3K9(组蛋白3末端第9位赖氨酸)、H3K14、H3K27、H3K36、H4K5、H4K8、H4K12和H4K16等,其中研究最充分的是H3K27。一般来说,H3K27ac主要位于活跃转录基因的启动子和增强子区域,在这些区域它与H3K4me3共存,一起促进基因激活表达。此外,H3K27ac还可以基因间区域形成超级增强子,进一步促进基因表达。
现研究表明,组蛋白乙酰化与植物的各种生命过程以及人类疾病的发生息息相关,如H3K9ac在杨树遭受干旱胁迫时发挥作用[5];H3K27ac在白血病中发挥重要作用[6]。目前,事实上,一些组蛋白乙酰化酶和脱乙酰化酶的激活剂和抑制剂已被批准用于治疗某些癌症的治疗。 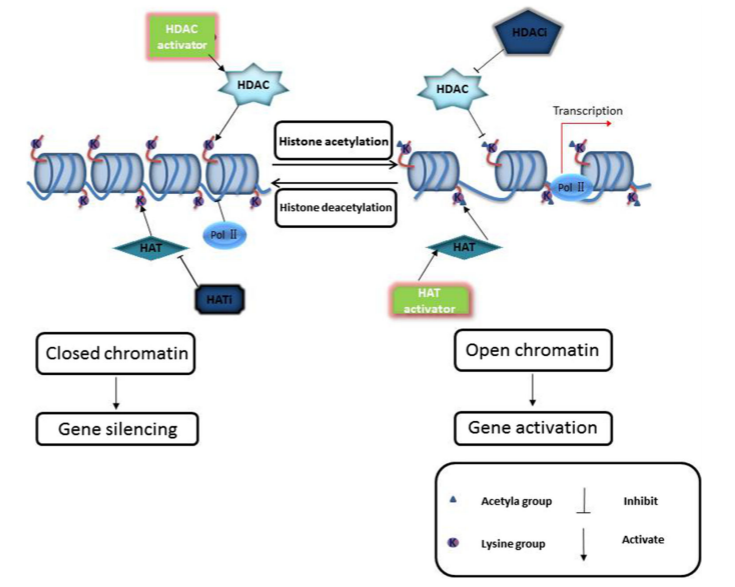 图3 组蛋白乙酰化与去乙酰化[7]
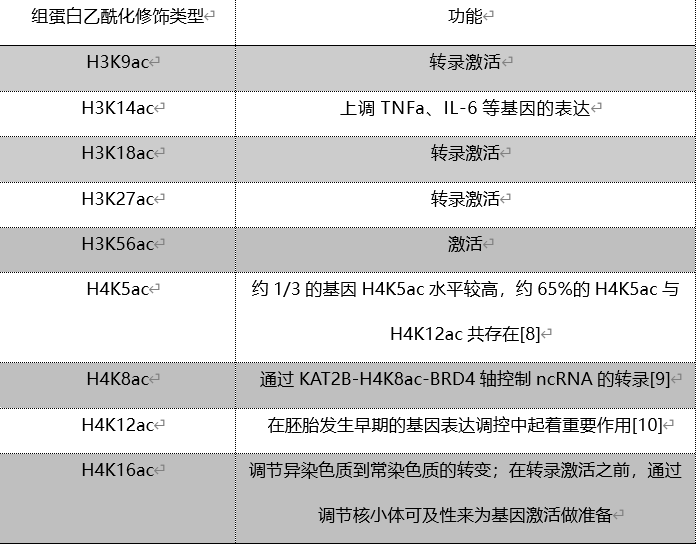 表1 部分组蛋白乙酰化位点及功能举例
3 组蛋白甲基化修饰 // 组蛋白甲基化主要发生在赖氨酸和精氨酸残基上,这也是一种重要的转录后修饰,是由组蛋白甲基转移酶催化完成的。其中赖氨酸残基可以发生单甲基化、二甲基化和三甲基化,分别用me1、me2和me3表示。组蛋白甲基化可以激活或者抑制转录。一般H3K4、H3K36和H3K79被认为是与转录激活有关,而H3K9、H3K27和H4K20被认为是与抑制转录有关[11]。但同一个位点甲基化基团的个数会影响具体的功能,如H3K27me1与激活有关,而H3K27me3则与抑制有关。多个研究表明,组蛋白甲基化在疾病发生过程中发挥重要作用,所以可以用于疾病诊断、预后以及药物研发。
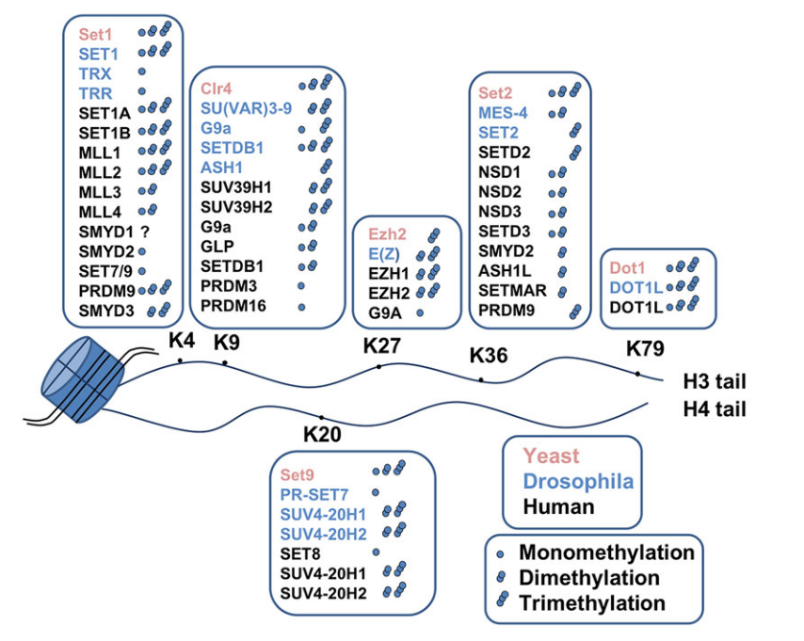 图4 组蛋白甲基转移酶及其在组蛋白上的赖氨酸靶向位点
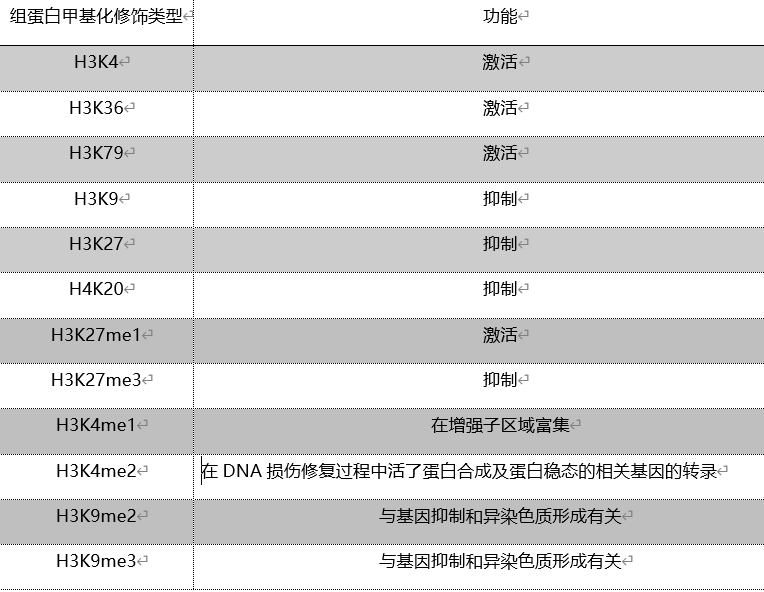 表2 部分组蛋白甲基化位点及功能介绍
4 组蛋白修饰研究方法举例 // 由此可见,了解组蛋白修饰并阐明其如何发挥作用对该领域的发展至关重要。近年来,组蛋白修饰方面的研究也与日俱增。在pubmed数据库中可以看到该方向的文章数量逐年增加,仅2021年全年发表的文章数就超过900篇。说明组蛋白修饰是目前的热门研究方向,而基础实验方法的发展也是促进该领域成果不断产出的关键所在。组蛋白修饰研究方法有western blot、质谱法以及基于二代测序的方法等,这里我们重点介绍常用的两种方法—Chromatin immunoprecipitation(ChIP)-Seq和Cleavage Under Targets and Tagmentation(CUT&Tag)法。
ChIP-Seq是2009年出现的、将ChIP(染色质免疫共沉淀)和NGS(二代测序)相结合的一种方法,该方法通过抗体免疫沉淀来分离蛋白的目标修饰及其结合的基因组DNA,并将相关DNA进行片段化及测序,以此来确定组蛋白修饰在基因组上的位置及丰度(图4)。该技术是研究组蛋白修饰在整个基因组中定位的金标准。
CUT&Tag是2019年发表的一种新方法,用于研究蛋白质与DNA的相互作用[12]。通过融合Tn5转位酶的抗体靶向标记捕获DNA-蛋白质的相互作用,在这个过程中把测序接头添加到靶序列上,后通过纯化DNA片段并进行测序来开展各种分析。
图4 组蛋白PTM研究方法举例[4] // 两种技术的相同之处在于,都通过特异性抗体捕获所要研究的组蛋白修饰,后分离与组蛋白修饰相结合的DNA,并通过对DNA进行测序和分析来推断组蛋白修饰的位置及丰度。但相比于ChIP-Seq,后者更有优势。CUT&Tag的优势体现在下面几点:首先,CUT&Tag不需要进行甲醛固定和超声打断;第二,需要的样本起始量更低;第三,因不需要固定,打断等操作,所以数据信噪比高,可重复性好。也正因为此,CUT&Tag正逐渐成为研究组蛋白修饰以及组蛋白与DNA相互作用的一种重要手段。
图5 两种技术流程比较
5 通过ChIP-Seq技术研究组蛋白乙酰化修饰的案例解析 为方便大家了解上述方法如何应用于研究中,这里小编也为大家附上一篇通过ChIP-Seq研究H3K27ac的案例供大家参考。
发表期刊:Nat Commun 影响因子:17.694 发表时间:2021年9月 研究背景:间变性大细胞淋巴瘤(ACLC)是一种侵袭性CD30阳性T细胞淋巴瘤T细胞淋巴瘤,包括全身间变性淋巴瘤激酶(ALK)阳性、ALK阴性原发性皮肤和乳房植入物相关的ALCL。一些ALCL亚组的预后仍不令人满意,并且缺乏有效的二线治疗选择。为确定可定义ALCL细胞状态和依赖性的基因,该研究通过全基因组范围内的H3K27ac ChIP-Seq来表征超增强子(SE)。 样本来源:ALCL、T细胞白血病衍生和HEK293T细胞系、FFEP样本等。 研究方法及技术路线: 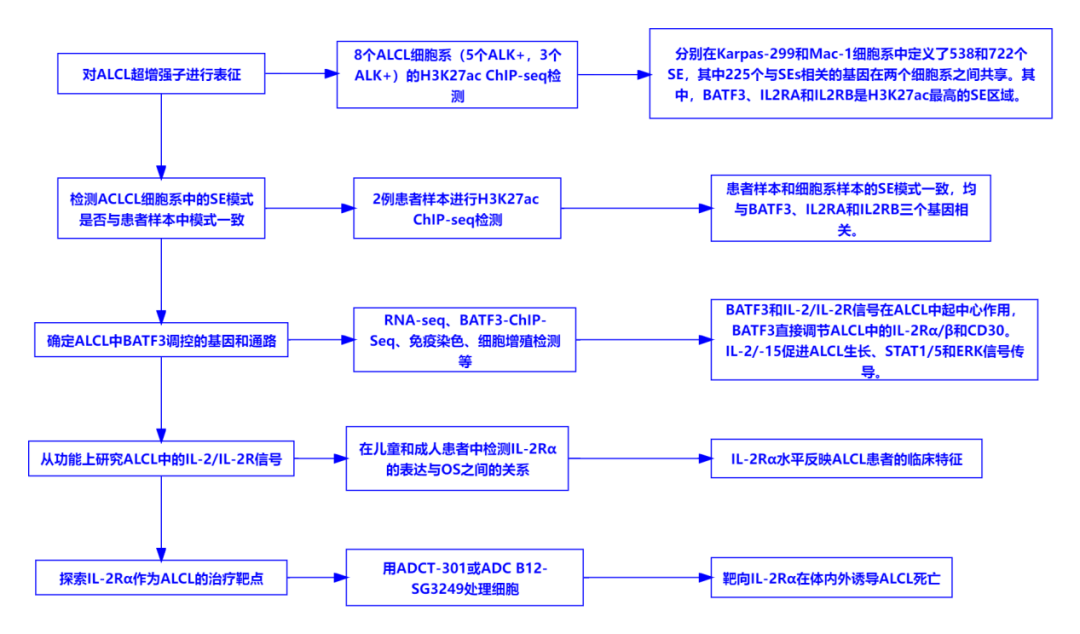 研究结论:该研究强调了BATF3/IL-2R在ALCL生物学中的重要性,并确定靶向IL-2Rα是一种有潜力的ALCL治疗策略。
研究点评:严格来讲,该研究使用了多组学技术联合策略。但主要基于H3K27ac ChIP-Seq的检测结果,再结合RNA-Seq以及基础细胞学实验进行后续分析,最终解析出具体调控机制以及可能的治疗靶点。 具备多种组学项目服务经验的伟寰生物,表观遗传方向更是我们的优势所在。不管是ChIP-Seq,还是CUT&Tag,都有提供。而且我们擅长处理组织样本,可以提供个性化1V1服务。如果您有任何需求,我们随时欢迎您的垂询!
参考文献
[1]Hiroshi Kimura, Histone modifications for human epigenome analysis. J Hum Genet. 2013 Jul;58(7):439-45. doi: 10.1038/jhg.2013.66. Epub 2013 Jun 6.
[2]Jose V Torres-Perez, Histone post-translational modifications as potential therapeutic targets for pain management. Trends Pharmacol Sci. 2021 Nov;42(11):897911.doi:10.1016/j.tips.2021.08.002. Epub 2021 Sep 23.
[3]Shuai Zhao, The language of chromatin modification in human cancers. Nat Rev Cancer. 2021 Jul;21(7):413-430. doi: 10.1038/s41568-021-00357-x. Epub 2021 May 17
[4]XueyuanLeng,AG(enomic)P(ositioning)S(ystem) for Plant RNAPII Transcription.TrendsPlantSci.2020Aug;25(8):744764.doi:10.1016/j.tplants.2020.03.005. Epub 2020 Apr 11.
[5]Verandra Kumar, Histone acetylation dynamics regulating plant development and stress responses. Cell Mol Life Sci. 2021 May;78(10):4467-4486. doi: 10.1007/s00018-021-03794-x. Epub 2021 Feb 27.
[6]Liling Wan, ENL links histone acetylation to oncogenic gene expression in acute myeloid leukaemia. Nature. 2017 Mar 9;543(7644):265-269. doi: 10.1038/nature21687. Epub 2017 Mar 1.
[7]Pingping Guo, et al. The Histone Acetylation Modifications of Breast Cancer and their Therapeutic Implications. Pathol Oncol Res. 2018 Oct;24(4):807-813. doi: 10.1007/s12253-018-0433-5. Epub 2018 Jun 11.
[8]C Sehwan Park, et al. Genome-wide analysis of H4K5 acetylation associated with fear memory in mice. BMC Genomics 2013, 14:539.
[9]Shuhei Ishikura, et al. ZFAT binds to centromeres to control noncoding RNA transcription through the KAT2B-H4K8ac-BRD4 axis. Nucleic Acids Res. 2020 Nov 4;48(19):10848-10866. doi: 10.1093/nar/gkaa815.
[10] K12 Markus Vieweg, et al. Methylation analysis of histone H4K12ac-associated promoters in sperm of healthy donors and subfertile patients. Clin Epigenetics. 2015 Mar 19;7(1):31. doi: 10.1186/s13148-015-0058-4. eCollection 2015.
[11] Yanjun Zhang, et al. Overview of Histone Modification. Adv Exp Med Biol. 2021;1283:1-16. doi: 10.1007/978-981-15-8104-5_1.
[12] Hatice S. Kaya-Okur, CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nat Commun. 2019; 10: 1930. Published online 2019 Apr 29. doi: 10.1038/s41467-019-09982-5.
[13] Huan-Chang Liang, et al. Super-enhancer-based identification of a BATF3/IL-2R-module reveals vulnerabilities in anaplastic large cell lymphoma. Nat Commun. 2021 Sep 22;12(1):5577. doi: 10.1038/s41467-021-25379-9.
|  /3
/3 