金桔
金币
威望
贡献
回帖0
精华
在线时间 小时
|
参考链接:https://www.youtube.com/watch?v=wIHwHDt2NoI
论文链接:https://www.biorxiv.org/content/10.1101/2022.12.09.519842v1.full.pdf
代码链接:https://github.com/RosettaCommons/RFdiffusion 背景
1. 介绍
蛋白质设计意义重大。目前深度学习方法主要在序列设计,脚手架功能位点设计,环低聚物设计以及抗体设计等多方面取得较大进展。但目前并没有提出一个通用的框架用来应对各类广泛的蛋白质设计任务。而且目前还没有技术能做到从头结合剂设计以及高阶对称环聚物的设计。
本文则提出了一个基于扩散模型的通用的蛋白质设计框架,结果显示该框架在各类任务中均优于已有的方法,且能实现从头结合剂设计以及高阶对称环聚物的设计。
序列设计:已知蛋白质骨架结构,设计出能折叠成该骨架的蛋白质序列。
脚手架功能位点设计:创建一个稳定的支架来支持目标基序。在这里,motif是具有生物学功能的结构蛋白片段,而scaffold则稳定了motif的结构
环低聚物设计:环状蛋白低聚物在几乎所有的生物过程中都起着关键作用,占蛋白质数据库(PDB)中所有沉积结构的近30%。可用于小分子结合和催化以及纳米笼装配的构建。这种环状组合蛋白可用于药物载体或者递送系统,例如纳米颗粒疫苗(组装蛋白可以携带抗原形成一个类似病毒颗粒的小球,从而呈现携带的抗原,激活体内的免疫反应)
抗体设计:根据抗原结构等信息设计抗体相关信息。
2. 一般的蛋白质设计流程:
蛋白质骨架设计->序列设计->模拟筛选->实验测定

对于蛋白质骨架设计分各种使用场景有不同的设计方式。最早的骨架设计是给定想要的二级结构,例如几条  螺旋,几条 螺旋,几条  折叠,然后根据能量(2022年中科大发表在nature上的一篇就是用这种方式进行设计的)或者从已有蛋白质数据库中采样得到可能的骨架结构。还有根据已有的功能位点进行inpainting或者幻化设计等,这两种方式可以根据已有的功能位点即得到生成后的结构也会得到生成后的序列信息。 折叠,然后根据能量(2022年中科大发表在nature上的一篇就是用这种方式进行设计的)或者从已有蛋白质数据库中采样得到可能的骨架结构。还有根据已有的功能位点进行inpainting或者幻化设计等,这两种方式可以根据已有的功能位点即得到生成后的结构也会得到生成后的序列信息。
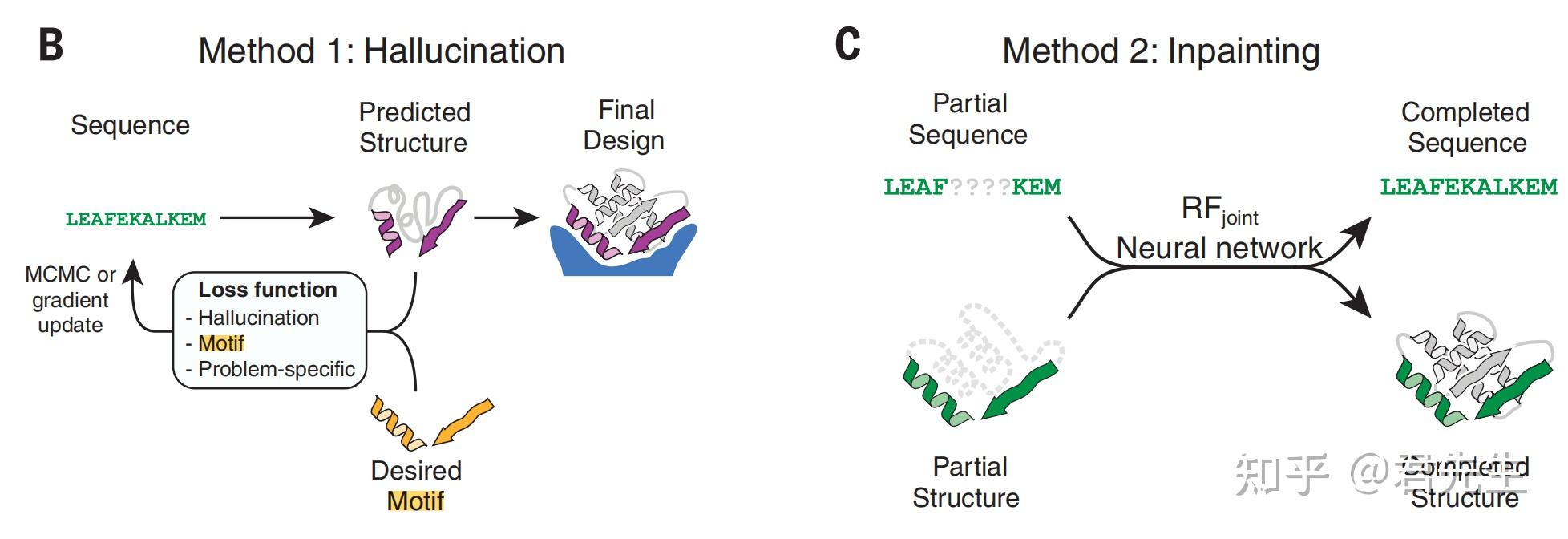
给定功能位点处的氨基酸类型以及结构,设计出能包含有该功能位点的整体蛋白质的结构。
根据motif进行蛋白质设计:

环状低聚物设计:
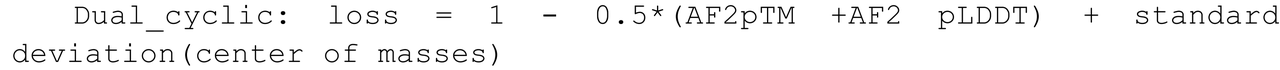
2. Inpating
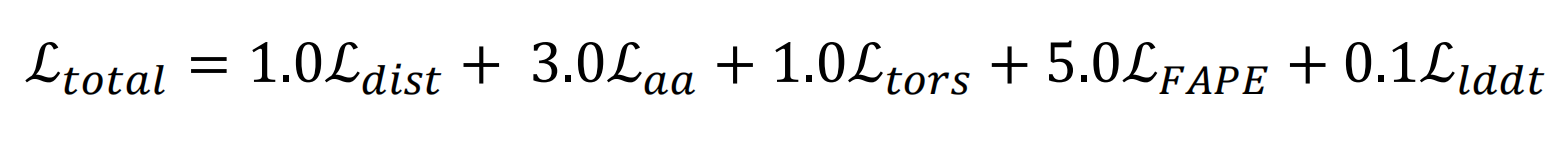
dist:距离损失,aa: 氨基酸类型的损失,tors: 扭转角的损失, FAPE: 结构坐标损失, lddt: 另一种结构损失。
以上所有结构损失应用在蛋白质的所有氨基酸上,只有序列损失应用在mask的氨基酸上。
3. RosettaFold2
RosettaFold的模型结构图
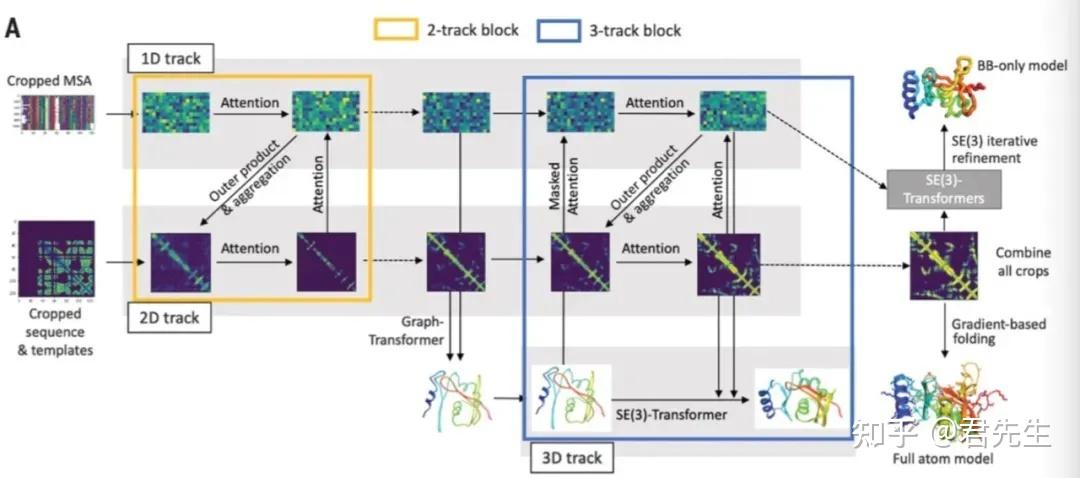
RosettaFold2的模型结构图
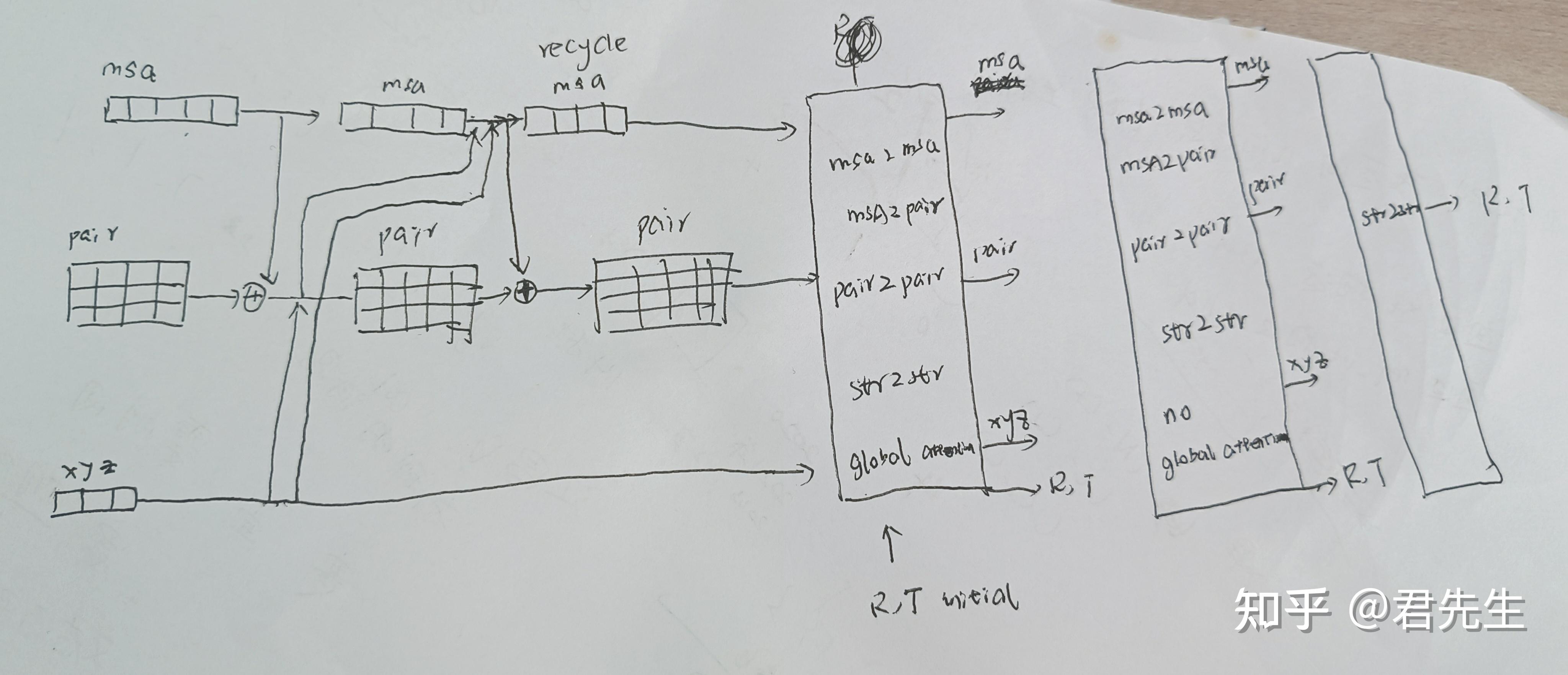
Rsettafold2的改进内容:
- 使用三重框架,其中3d信息的初始化来自模板结构。
- 根据3d信息更新pair信息。
- 在1d,2d,3d增加attention交互信息。
- 增加recycle操作,不断更新input embedding。
正文
在此默认大家已经掌握扩散模型相关知识。
1. RFdiffusion的扩散框架介绍
RFdiffusion的目的是为了生成蛋白质骨架结构,所以本文对于骨架结构的生成看成是每个氨基酸局部坐标系相对于全局坐标系而言的旋转矩阵R和平移向量Z的生成。因为氨基酸局部坐标系一般都是以  原子为坐标原点,所以平移向量Z相当于就是 原子为坐标原点,所以平移向量Z相当于就是  原子在全局坐标系下的坐标。 原子在全局坐标系下的坐标。
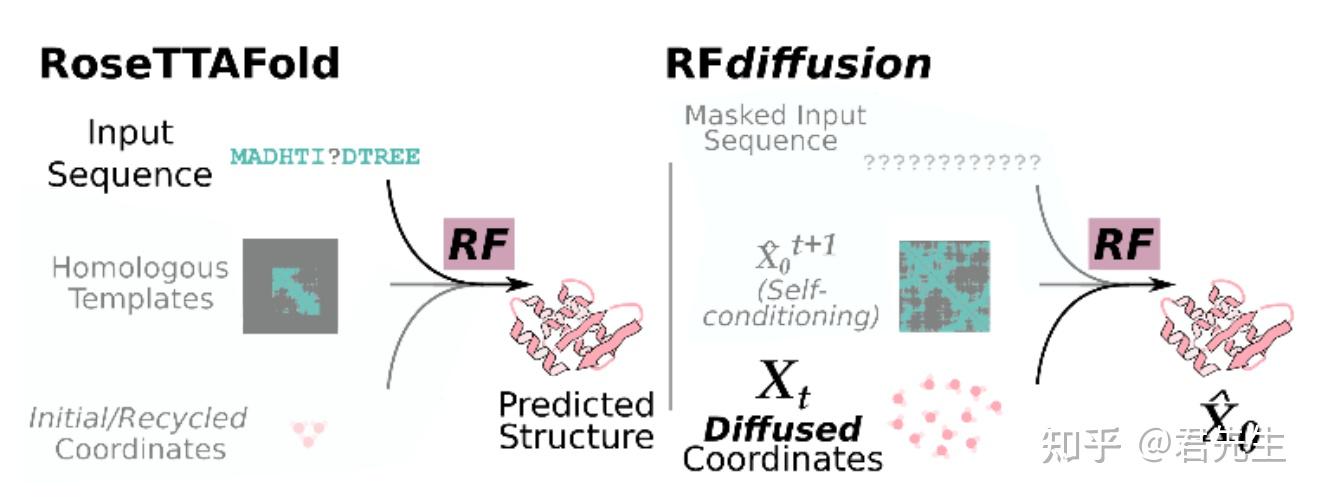
1. 预测过程
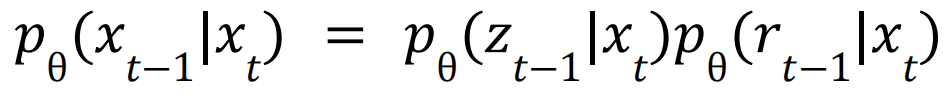
-公式1
作者近似的将旋转和平移视为独立分布的变量,并用实验证明了这样效果是最好的。
在使用公式1时,需要注意扩散模型需要保证公式2,即在全局坐标系发生旋转和平移时,预测的结果也应当发生相同的旋转和平移。其中 
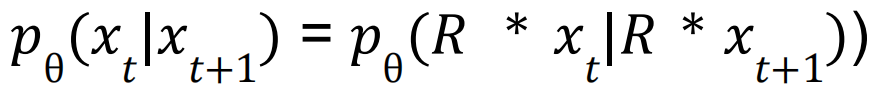
-公式2
2. 加噪去噪过程
平移向量加噪:直接在每一步扩散过程中引入高斯分布噪声,和普通的DDPM别无二致。
加噪:
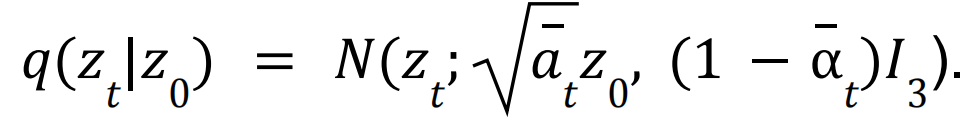
平移向量去噪:
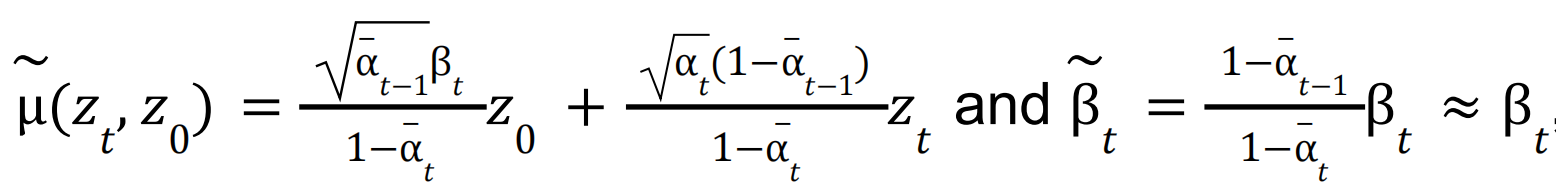
旋转矩阵加噪:特殊加噪,因为SO(3))是一个紧化的黎曼流形,其上的典型高斯分布不明确,所以之前推导的高斯分布下推导的各类式子不能适用。这里将生成建模扩展到黎曼流形上,并将扩散过程定义为在流形上的布朗运动。
加噪:
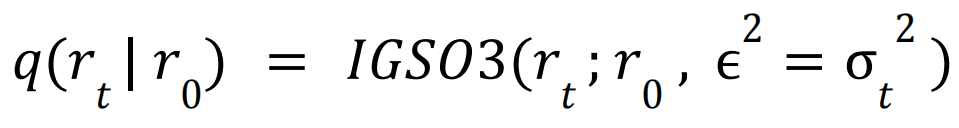
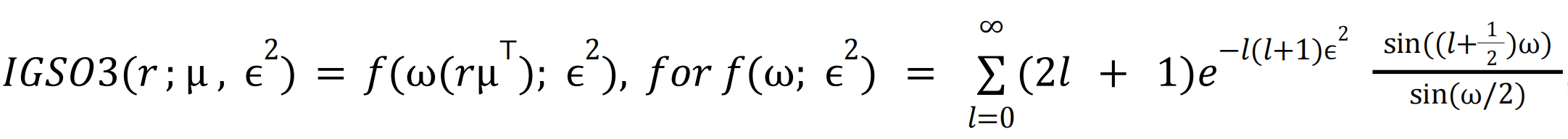
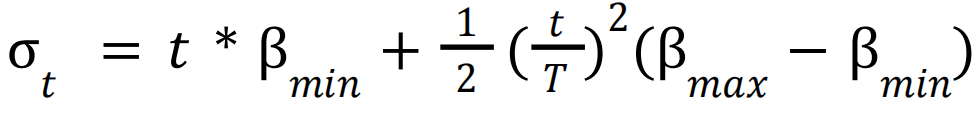
旋转矩阵去噪:
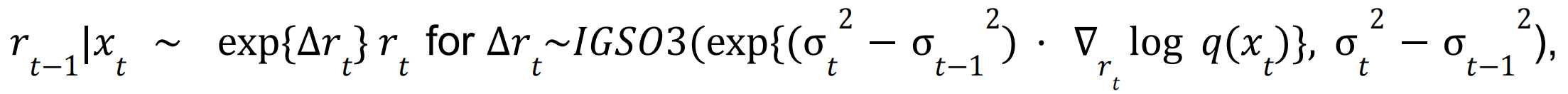
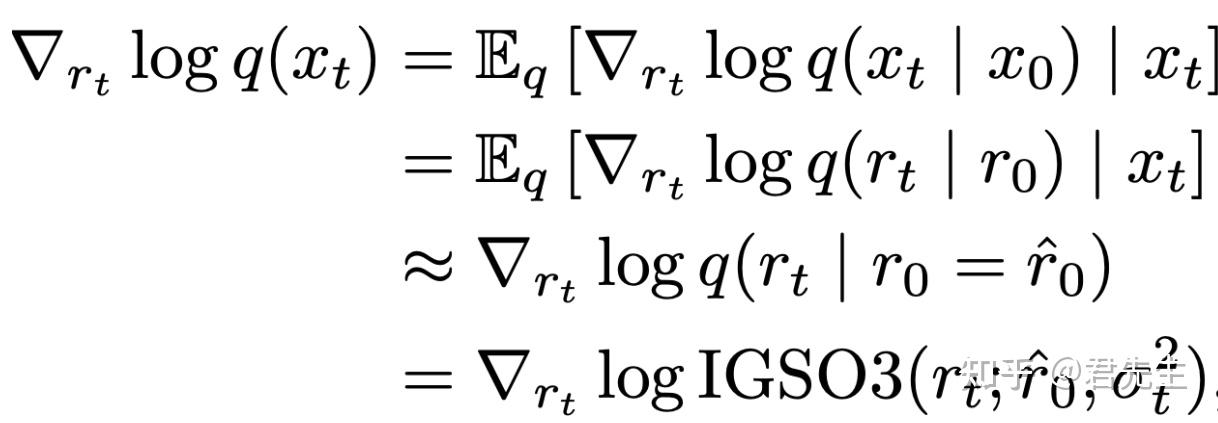
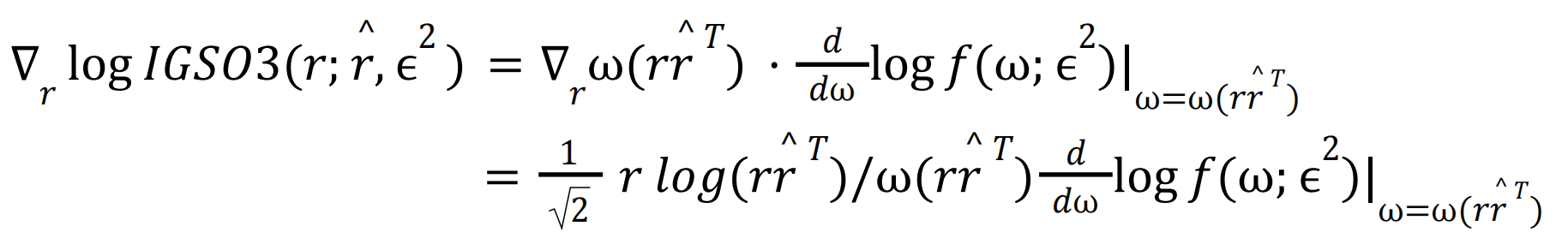
3. loss计算
平移损失:
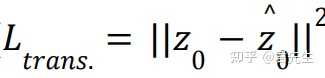
旋转损失:个人的一些理解:根据代数几何可知,下试减号右侧相当于将1向量先旋转r矩阵,然后再旋转r'^T,如果r'和r不一致,则旋转后的向量将会和1向量之间有夹角,否则应当重合。这里衡量了旋转后两向量之间的欧氏距离。
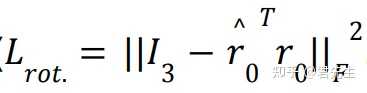
4. Self-Condition
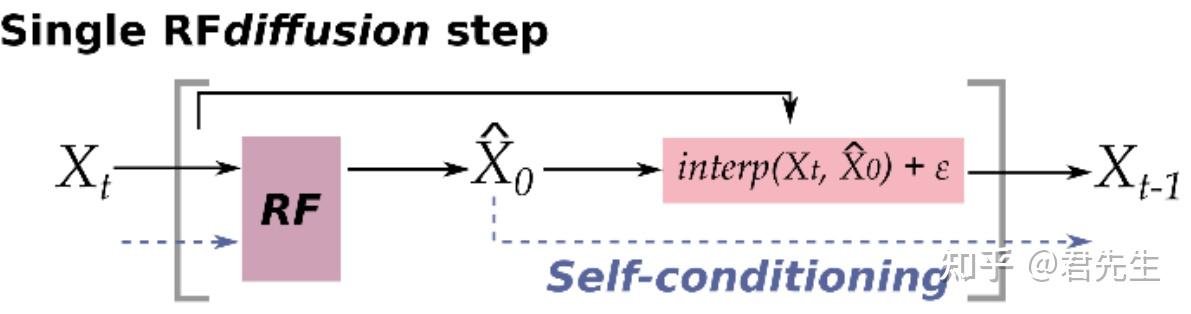
是模型训练过程中的一个trick,如下所示,其中50%的样本走正常流程,即根据  预测的 预测的  计算loss。剩下50%的样本会先通过 计算loss。剩下50%的样本会先通过  预 预  ,然后根据 ,然后根据  预测 预测  ,再通过 ,再通过  以及 以及  ,预测更新一层的 ,预测更新一层的  ,从而形成 ,从而形成  的自循环。 的自循环。

5. 扩散模型伪代码



2. 无条件蛋白质单体生成
先对RosettaFold2进行训练,之后载入训练好的RosettaFold2的模型再在单体数据集上进行无条件结构生成训练。
1. RosettaFold2的训练
- 数据集:PDB库中单体/低聚物数据;异质多聚物数据;Alphafold预测pLDDT > 0.7的数据;随机采样的负bind样本。比例为:2:1:4:1
- loss组成:(MLM) loss, distogram (dist) prediction loss, FAPE loss, accuracy estimation loss, bond geometry loss and van der Waals (vdW) energy loss
2. RFdiffusion Training
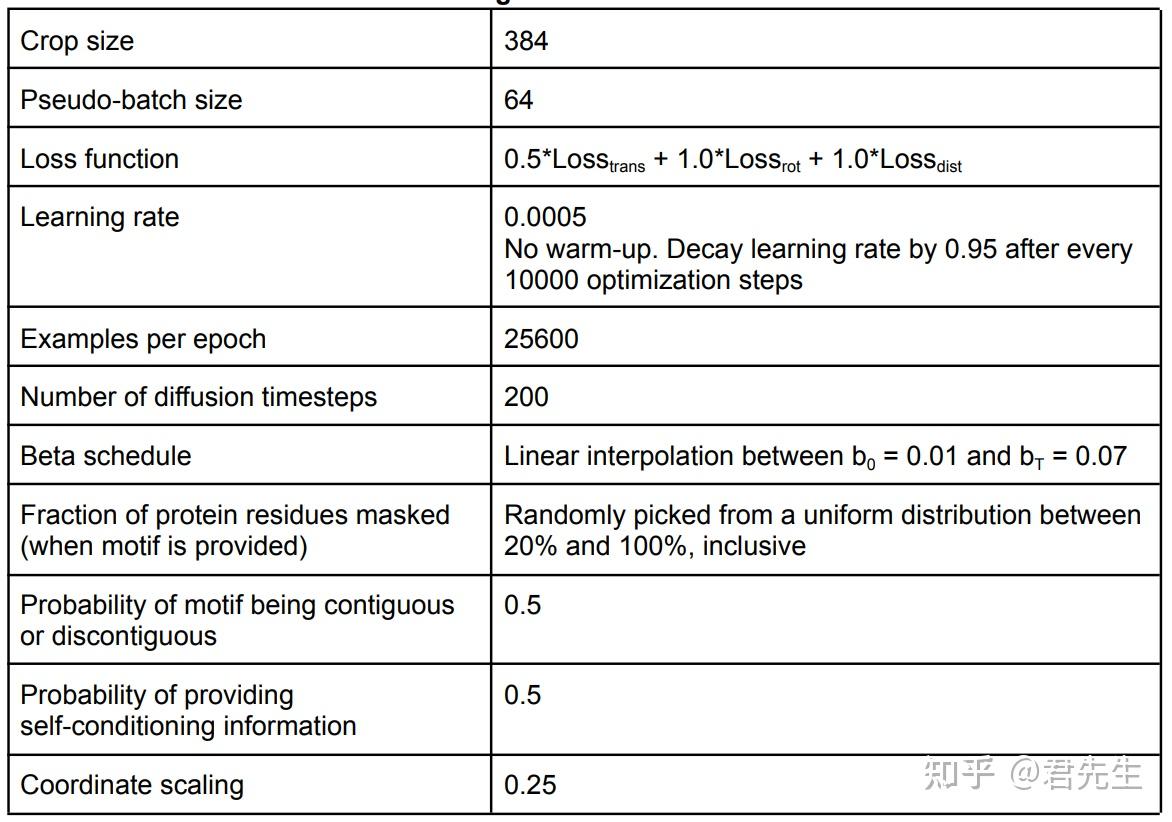
在单体数据集上训练,具体训练超参如下:RFdiffusion trains to convergence when initialized from RF2 weights in ~5 epochs
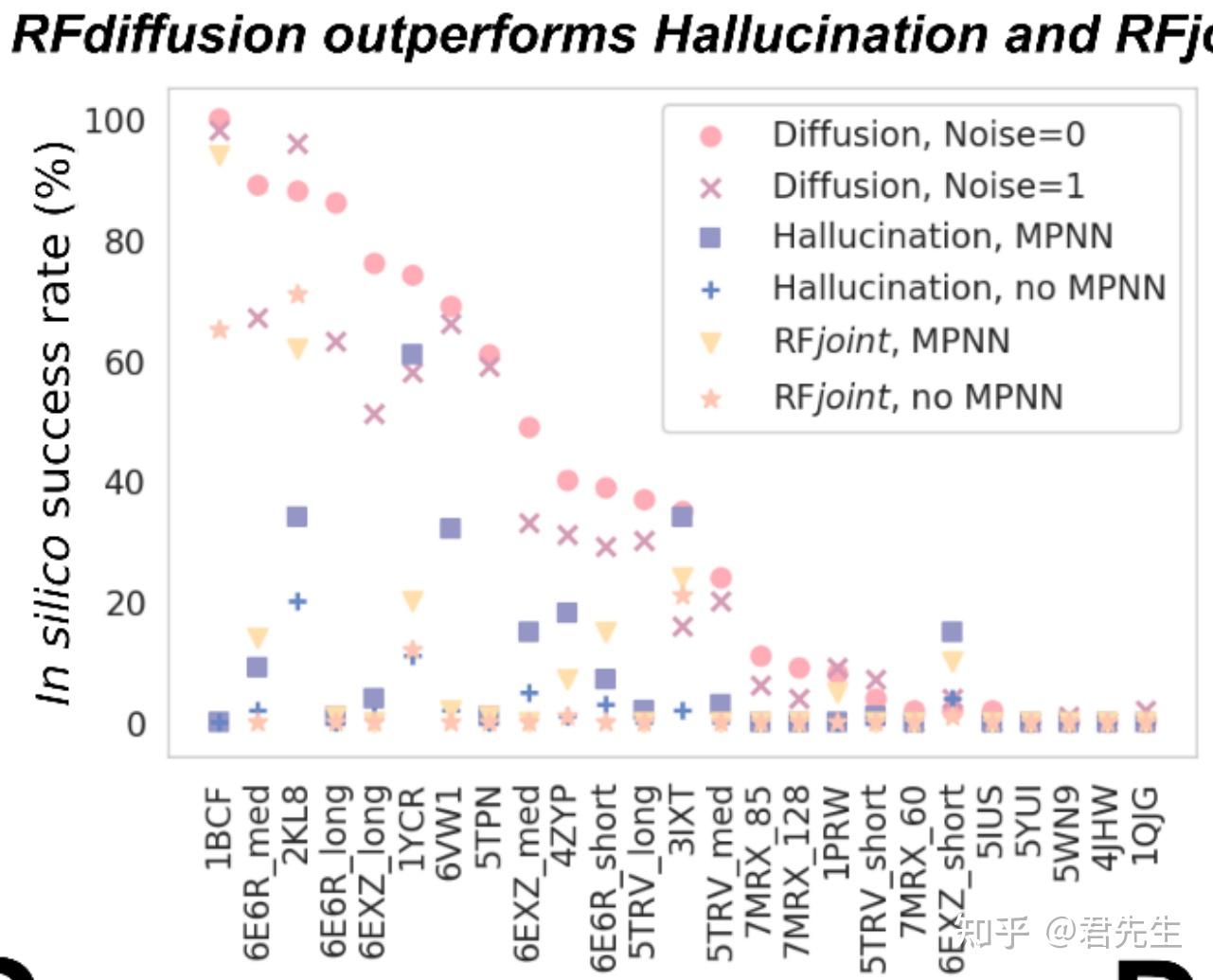
3. 结果
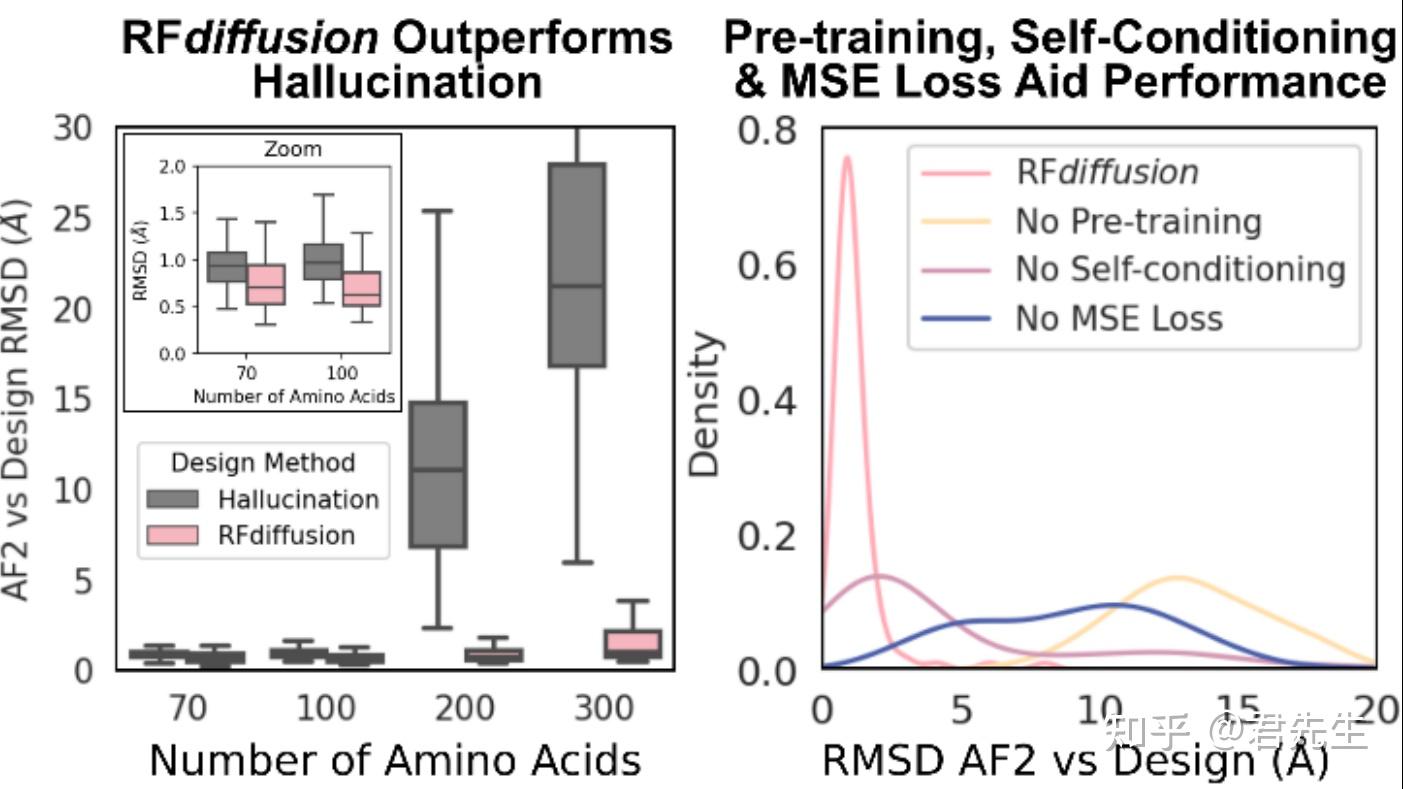
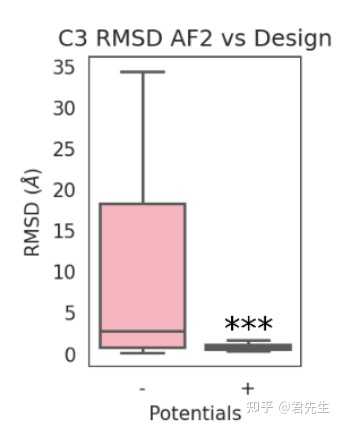
左图显示,RFdiffusion和hallucination相比,性能要高出很多。
右图显示,RFdiffusion在训练时的各种策略对结果的影响。
- 当去掉预训练时,性能下降很多。
- 去掉self-condition时,性能下降很多。
- 去掉mse loss,将mse loss替换为FAPEloss时,也下降很多。
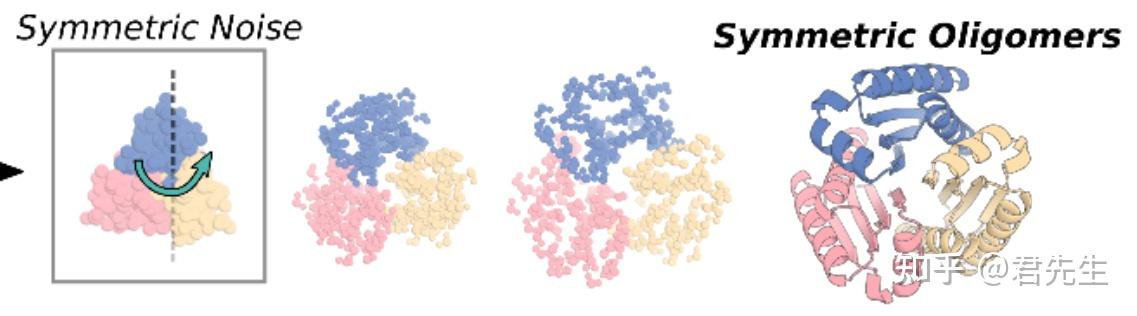
3. 通过显式对称去噪的高阶低聚物设计
1. 功能描述
设计出一些满足对称结构的环状低聚物。该低聚物可以用来做疫苗平台,药物运输和催化剂。
在给定单体条数,以及单体序列长度后,通过RFdiffusion设计出可行的对称环状低聚物。
2. 实现方式
点群对称性可以用旋转矩阵的有限集合来表示,我们可以用围绕z轴旋转(360/K)°的旋转矩阵集合来表示K阶循环对称组
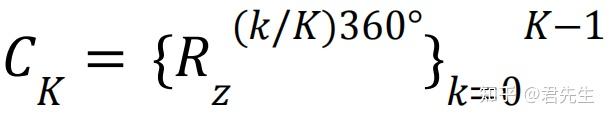
即对于一个多聚体  . .
对于其中的某一个单体 
则只要重建出  ,完整的多聚体如下所示: ,完整的多聚体如下所示:
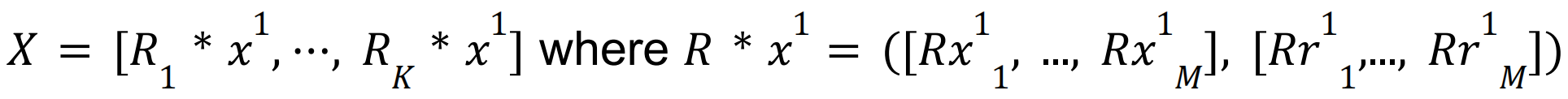
重建  则利用无条件生成的方式进行重建即可。 则利用无条件生成的方式进行重建即可。
与之前的幻化方式相比,提高了对称的精确性,并且节省了内存和计算。
3. 结果
A图显示不同情况下设计的堆成环聚物的结构展示。
B,C图从左到右依次为RFdiffusion预测的结构;AF2通过proteinmpnn预测的序列,再预测结构得到的示意图;电镜下2D类平均值;电镜下的3D示意图。从这些图中可以看出实际结构与设计非常接近。
E图:二十面体有60个亚基排列在2重、3重和5重对称轴周围。在被选中进行实验验证的48个二十面体中,有一个被nsEM确认形成预期的装配。像HE0902这样的设计(以及未来类似的大型组装)应该可以作为新型纳米材料和疫苗支架,具有强大的组装能力
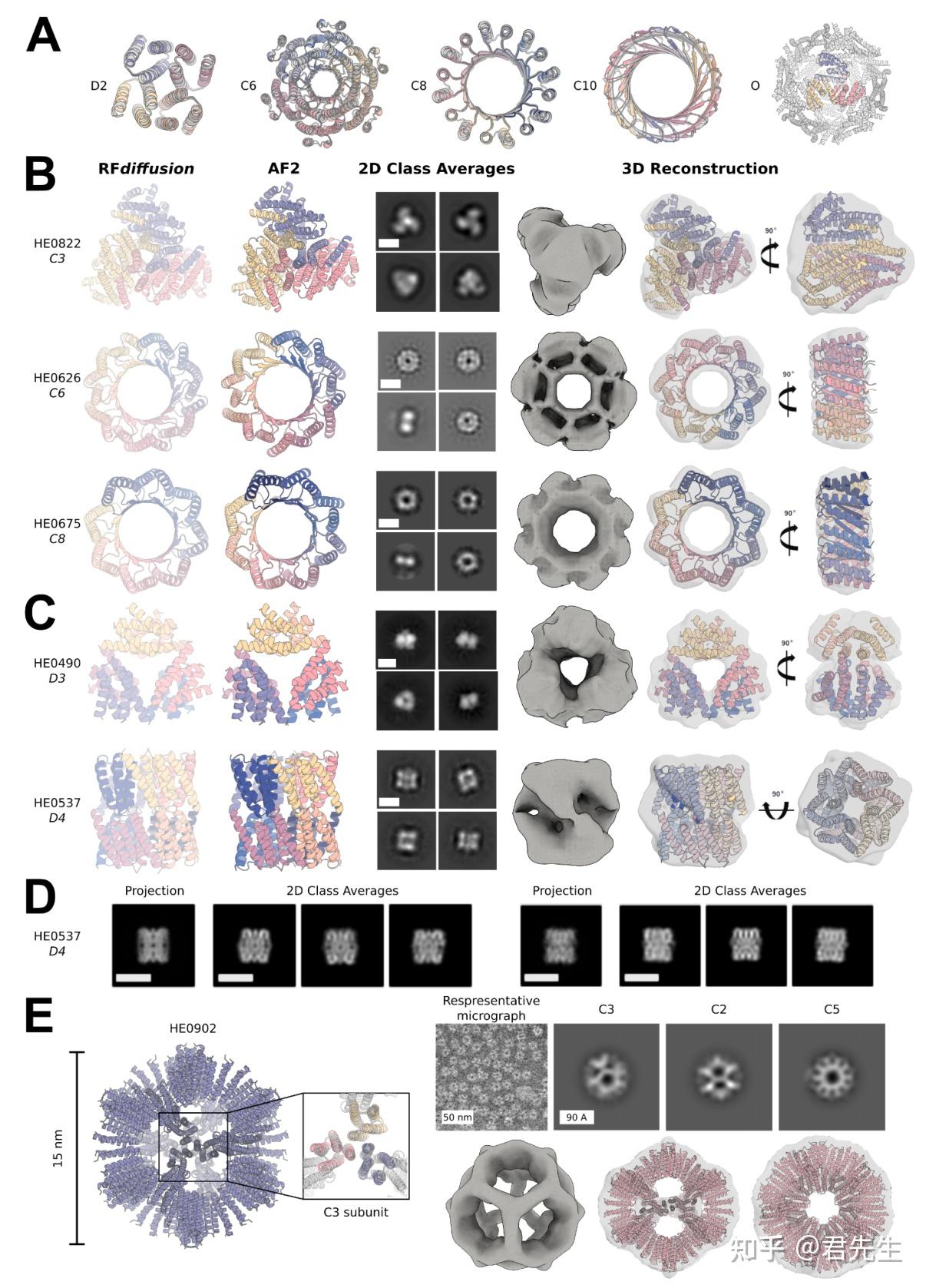
在辅助链间和链内接触电位的引导下(看第7节内容)对性能的影响
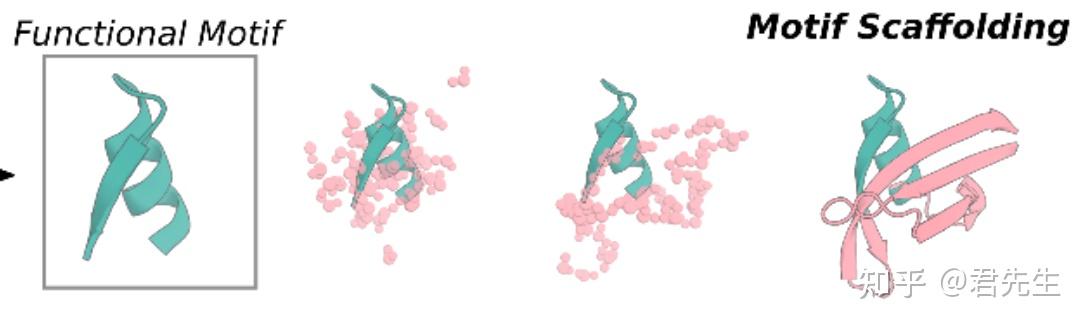
4. 包含功能位点的脚手架设计
1. 功能描述
已知某段连续或者不连续的功能位点的氨基酸信息以及结构信息,创建一个稳定的支架来支持功能位点。如下图所示。
2. 实现方式
设功能位点的坐标为  ,骨架坐标 ,骨架坐标  ,则最终要构建的完整的坐标为 ,则最终要构建的完整的坐标为  。 。
目标是:
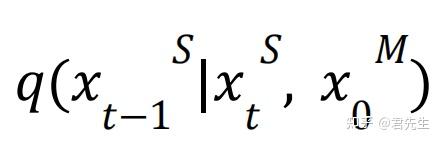
3. 训练方式
- 从pdb数据库中随机选择一
 . .
- 将
 序列随机分割为 序列随机分割为  ,遵循RF diffusion . ,遵循RF diffusion .
- 给骨架坐标上加噪。
- 计算出在给定
 时的loss. 时的loss.
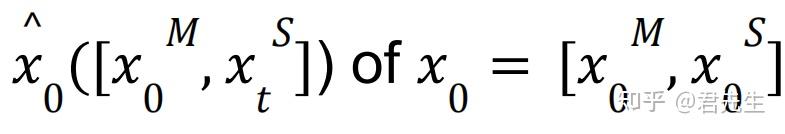
为了鼓励功能位点上的结构信息不发生改变,所以我们在预测$$$$x_0^S $$$$时,每一步去噪中用的功能位点的输入坐标信息即为原始坐标信息,并且还会引入功能位点的侧链扭转角以及氨基酸序列信息。并同时计算在功能位点和骨架上的预测损失。
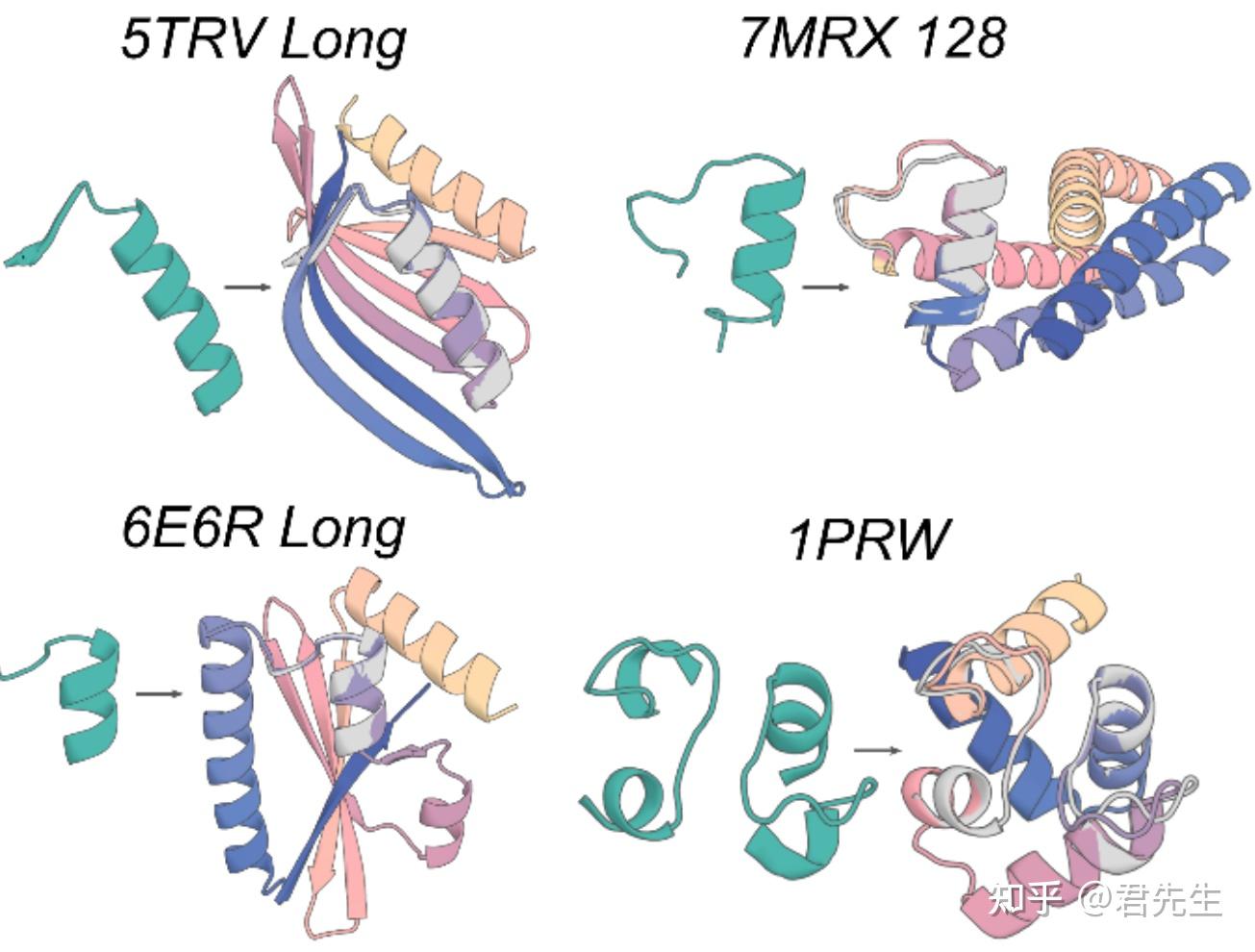
4. 结果
绿色部分为已知的要包含的功能位点,彩色的为 AF2 prediction of an RFdiffusion design。
5. 脚手架酶活性位点
1. 功能描述
蛋白质设计中的一个重大挑战是构建出包含少量单氨基酸的酶活性位点的骨架设计。RFdiffusion能够在一系列酶类中以高准确率获得包含多个侧链和主干功能基团的支架酶活性位点。
实现方式
- 对酶活性位点支架进行微调的RFdiffusion版本是从训练了5个epoch的基本版本(uncondition)开始训练的
- 在微调过程中,30%的任务来自基本模型任务集,其他70%是三重接触任务,其中随机选择3个残基,这三个残基的要求是:与该残基Cβ-Cβ距离小于6Å的残基数量大于10个残基。则这样的残基可被选择为具有活性位点的残基。最后随机选中的这三个残基作为功能位点进行后续的骨架的生成。
- 选中的残基会有50%的概率保留侧链信息。
- 加大对于功能位点处位移损失的权重,这个权重设置为10。在这里加大权重的原因是,该任务的功能位点长度过短,所以需要尽可能保证在功能位点处的不变性。
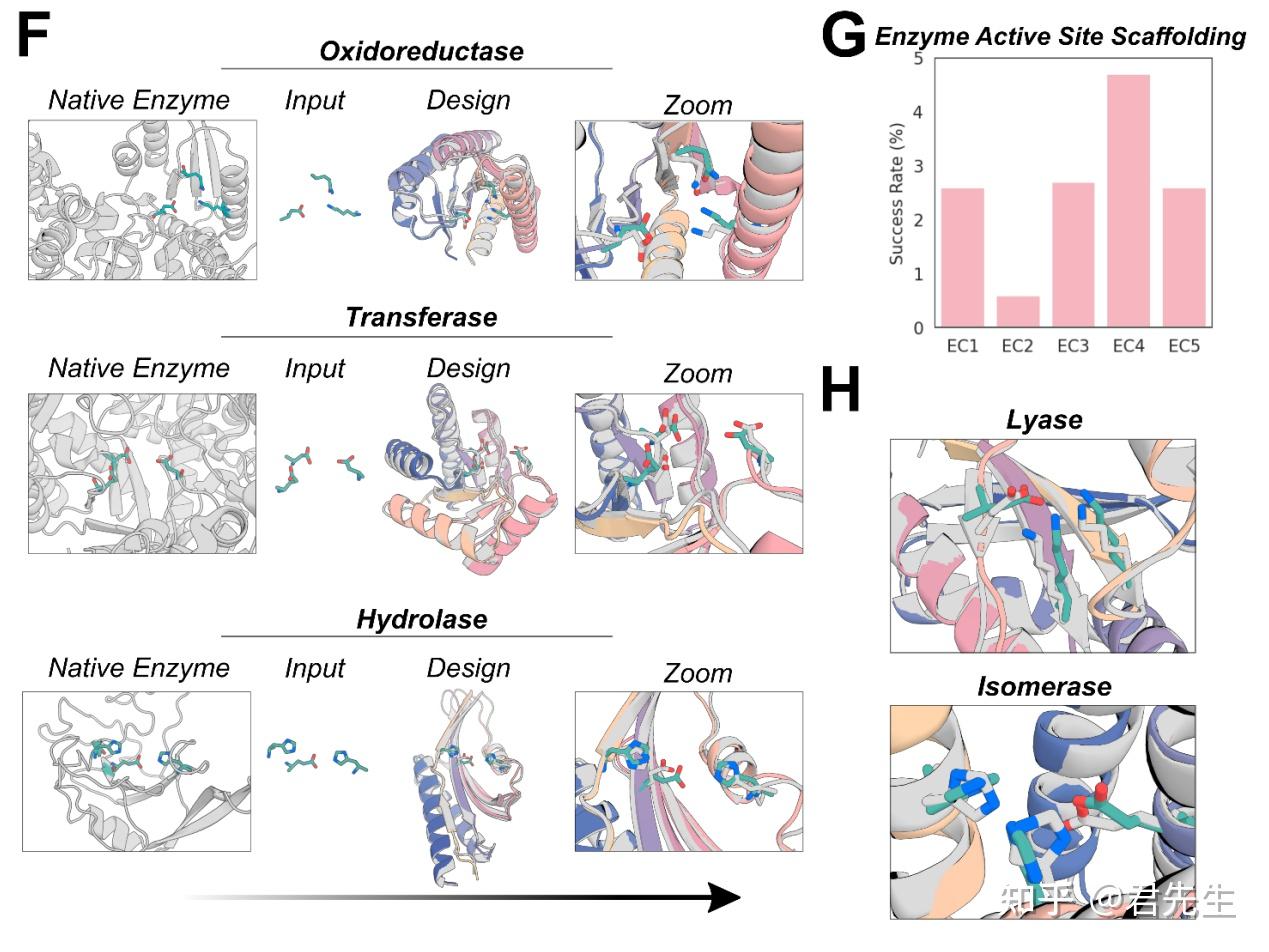
2. 结果
如下所示为不同酶的类型下生成的产物的展示。
其中G图展示的是在不同酶类型下,设计的成功率的占比。成功的设定规则为:AF2 Motif RMSD vs native: backbone < 1Å, backbone and sidechain atoms < 1.5Å, RMSD AF2 vs design < 2, AF2 pAE < 5
6. 全新蛋白和肽结合剂设计
1. 功能描述
设计靶向蛋白的高亲和力结合物是蛋白质设计中的一个巨大挑战,具有许多治疗应用。最近描述了一种基于物理的Rosetta方法,仅从目标结构信息重新设计结合剂到蛋白质结合剂的通用方法。随后发现利用ProteinMPNN进行序列设计,利用AF2进行设计滤波,提高了设计成功率。然而,实验成功率很低,每次设计活动都需要数千种设计进行筛选,而且这种方法依赖于预先指定一组特定的蛋白质支架作为设计的基础,这内在地限制了可能解决方案的多样性和形状的互补性。据我们所知,还没有一种深度学习方法在设计完全从头开始的粘合剂方面显示出实验上的普遍成功。
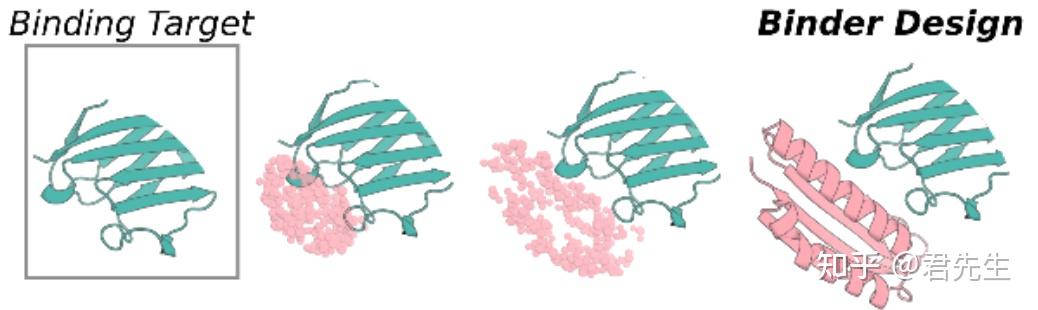
2. 实现方式
- 从base model(uncondition)上接着训练.
- 其中50%的数据来自复合物,50%的数据来自单体数据。
- 当数据为复合物数据时,只对其中一条链进行加噪,另一条链保持固定。并且还会提供hotspot residues的索引。hotspot residues的选取为与另一条链 Cβ-Cβ的距离包含在10Å以内。
- 在训练时,50%的时间会引入加噪对应链的二级结构信息。剩下50%的时间会引入块邻信息。
- 在训练时,会对二级结构信息或者块邻信息进行mask(0-75%)。以便在预测时,提供不够精确的二级结构或者块邻信息。
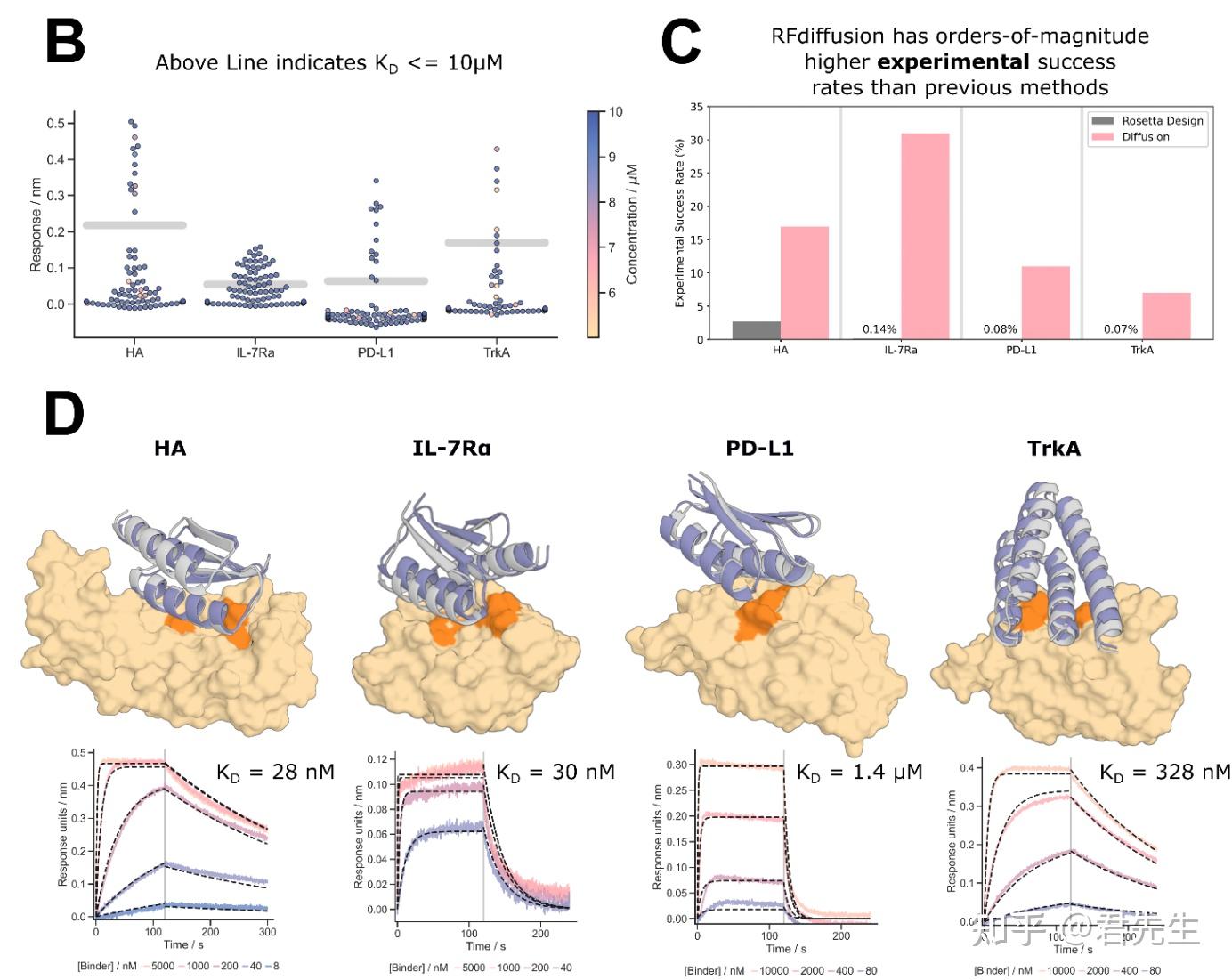
3. 结果
B图:在浓度  时,如果响应大于K_d = 10 μm时的响应时,认为具有更高的亲和力。 时,如果响应大于K_d = 10 μm时的响应时,认为具有更高的亲和力。
C图:和以往的方式相比,该文的方法比已有的方法的实验成功率要高很多。
D图:不同靶点下,RFdiffusion设计的结果展示。
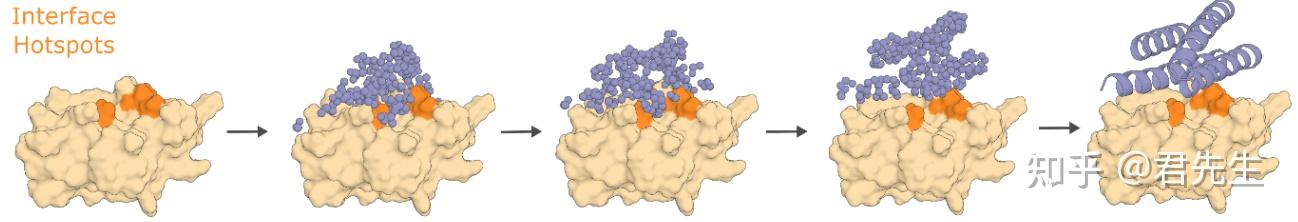
7. 用外部电位引导RFdiffusion推断
对于对称环状低聚物进行设计时,除了进行单体生成外,还希望借助外部点位引导最终的低聚物的设计。该过程只作用在预测过程。
整体思想是,根据扩散模型中的classifer guidance的条件引导思想。在进行去噪时,除了沿着数据分布方向进行采样外,还需要沿着向目标类型靠近的方向进行移动。
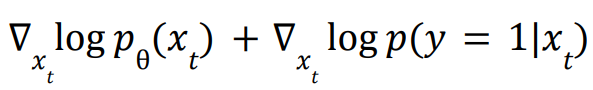
在本文中,也构造一种想要的目标,最后沿着该目标的方向进行采样生成。
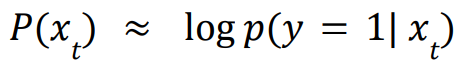
对于环状低聚物设计的额外目标可以用下列式子表示:如下所示,在下列式子的约束下,会优先形成亚单位之间的联系,同时鼓励单个亚单位能够很好的包裹在一起。
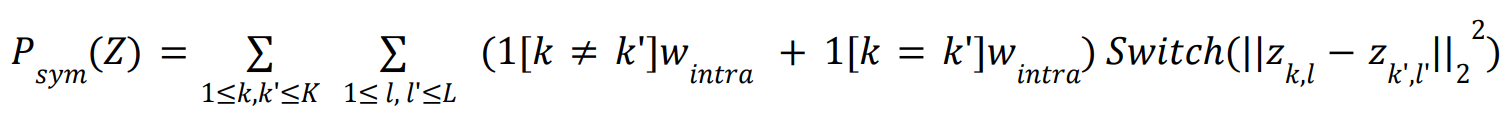
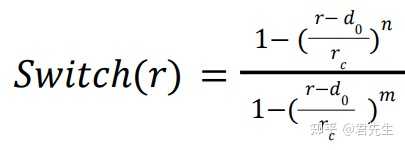
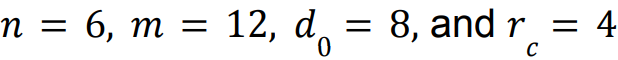
因为在高t点上矫正分布偏差足以促进高阶结构中的接触,而在低t点上没有必要继续这样做,因为四元结构已经充分确定,因此我们用guide-scale ( )来衡量外部电力引导
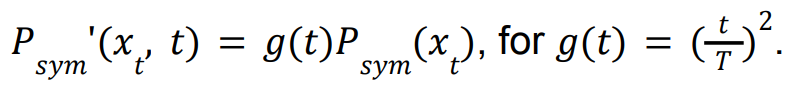
8. 总结
- 对结构进行扩散时,不是简单的对坐标进行扩散,而是对坐标以及旋转矩阵以不同的方式分别进行扩散。
- 对模型进行预训练,则给扩散模型带来很大的收益。并且扩散中使用的模型结构,信息的流动方式和预训练相差不大。只在扩散中多增加了时间t这个尺度。
- 在训练时,使用selfcondition策略提升模型性能。
- 在训练时,采用mse loss比fape loss效果更好。
- 对于各种下游任务进行巧妙的训练数据集的构造:酶活性位点,功能位点数据的构造,对接数据是否也能设计出训练数据集。
- 在进行蛋白质结合剂设计的时候,通过在二维信息上的mask策略,使得最终在结合剂设计时,可以依据想要的二级结构等信息设计出理想的结合剂。我们在进行其他任务设计时,或许也可以将条件刻画为一些基础数据集共有的特征,之后将这个特征进行一些mask.达到在预测时,依据设定特征进行设计的目的。
- 在进行条件引导时,可增加一些额外的目标函数,使得最终采样样本沿着额外目标函数的方向生成。就像用外部电位引导RFdiffusion推断那样。
|
|
 /3
/3 